Rosyjscy naukowcy odkryli nieznanego wcześniej bakteriofaga Sxt1 w terapeutycznym koktajlu fagowym do leczenia infekcji bakteryjnych. Jest on zdolny do infekowania wielu dzikich szczepów E. coli opornych na fagi z tej samej rodziny. Po porównaniu Sxt1 z pokrewnymi fagami T3 i T7, naukowcy odkryli, że sekret jego skuteczności tkwi w strukturze włókien ogonka. Wyniki badania opublikowano w czasopiśmie „Viruses”.
Jednym z rozwiązań problemu oporności bakterii na antybiotyki jest leczenie zakażeń bakteryjnych za pomocą bakteriofagów – wirusów infekujących bakterie. Fagi są jednak wysoce specyficzne i mogą infekować tylko określone szczepy jednego gatunku bakterii. Dlatego w terapii stosuje się koktajle fagowe – mieszaniny różnych fagów, które działają na różne szczepy jednego lub więcej gatunków bakterii. Jednak ze względu na brak danych dotyczących skuteczności klinicznej terapeutycznych koktajli fagowych, ich zastosowanie jest ograniczone.
Zespół naukowców z Laboratorium Analizy Metagenomu Skoltech pod kierownictwem Artema Isajewa zbadał skład terapeutycznego koktajlu fagowego „Sextaphage (poliwalentny piobakteriofag)”, produkowanego przez rosyjską firmę Mikrogen, i wyizolował nowego bakteriofaga, Sxt1, który był w stanie zakażać szeroką gamę szczepów E. coli, w tym te, które są oporne na inne, spokrewnione fagi.
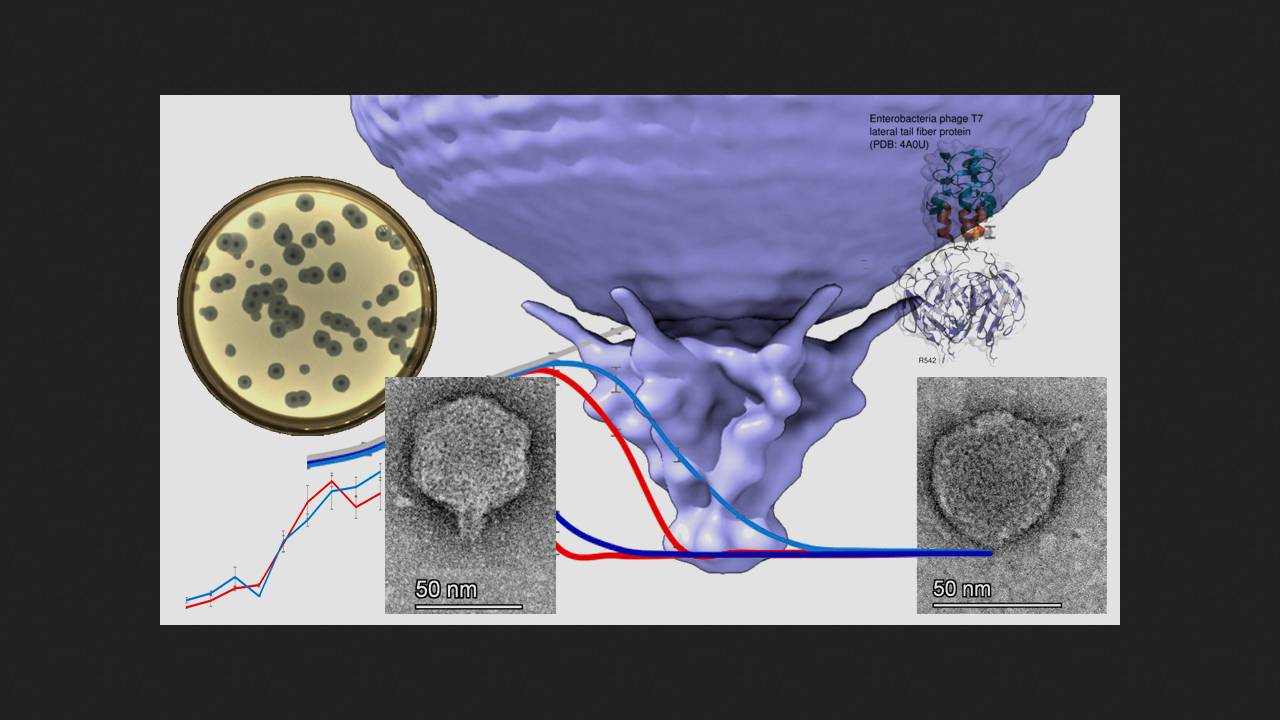
Koktajl polifagowy „Sextaphage” zawiera fagi wyizolowane ze Staphylococcus spp., Streptococcus spp., Proteus (P. vulgaris, P. mirabilis), P. aeruginosa, Klebsiella pneumoniae oraz enteropatogennej E. coli. W badaniu naukowcy wykorzystali szczep E. coli BW25113 jako gospodarza. Z koktajlu wyizolowano faga Sxt1 z rodziny Autographiviridae. Okazało się, że jest on bliskim krewnym fagów E. coli T3 i T7. Fagi z tej rodziny posiadają polimerazę RNA, która transkrybuje szereg genów fagowych. Geny wczesne (transkrypowane natychmiast po zakażeniu komórki bakteryjnej) Sxt1, geny wczesne (zaangażowane w metabolizm DNA i replikację genomu faga) oraz niektóre geny późne (kodujące składniki strukturalne wirionów fagowych, ich pakowanie i lizę bakterii) były niemal identyczne z genami faga T3. Różnice między Sxt1 a pokrewnymi fagami stwierdzono w genach kodujących włókna ogonkowe oraz dwa białka wewnętrzne i, według naukowców, są one wynikiem rekombinacji faga, prawdopodobnie z fagiem Berlinvirus.
Исследователи построили филогенетическое древо бактериофагов подсемейства Studiervirinae, куда входит Sxt1, определили его предполагаемое происхождение и пришли к выводу, что секрет его исключительности кроется именно в белке латеральных хвостовых фибрилл. Фаги из семейства Autographiviridae для прикрепления к бактериальной клетке используют шесть хвостовых фибрилл, каждая из которых состоит из тримера этого белка. На концах тримера имеются участки, контактирующие с поверхностью клетки. N-концевой домен необходим для прикрепления к капсиду, длинный пирамидальный домен включает в себя бета-листы от каждой из трех молекул, входящих в состав тримера, за ним расположен короткий альфа-спиральный линкер и концевой домен, отвечающий за взаимодействие с бактериальной клеткой. Пирамидальный домен Sxt1 содержал шесть вставок длиной более пяти аминокислот, причем некоторые из вставок образовывали дополнительные бета-слои. Линкер также включал в себя дополнительную вставку из 27 аминокислот. В итоге за счет вставок хвостовая фибрилла Sxt1 длиннее, чем у Т3 и Т7. С помощью программы AlphaFold2 ученым удалось смоделировать концевой домен Sxt1 с альфа-спиральным линкером и часть пирамидального, и оказалось, что она только на 43 процента идентична аналогичной области фага Т3 и на 54 процента — фага Т7. При этом хвостовые фибриллы Sxt1 распознают иной набор рецепторов бактериальной клетки по сравнению с Т3 и Т7.
Для оценки специфичности нового фага исследователи высеяли фаги Sxt1, T7 и T3 на распространенные штаммы E. coli: BW25113, MG1655, BL-21, B, C, DH5α, HS и Nissle1917, а также на штаммы F+ BW39773 и KD263, которые способны ингибировать T7 из-за наличия системы абортивной инфекции PifA. Устойчивыми ко всем фагам оказались только штаммы HS и Nissle1917. Все остальные штаммы E. coli фаг Sxt1 успешно инфицировал. При этом Т3 не смог заразить штаммы линии К12, а Т7 потерпел неудачу со штаммами с системой PifA.
Aby określić zakres gospodarzy faga Sxt1, naukowcy przetestowali jego interakcje z różnymi szczepami E. coli. Bakterie E. coli dzielą się na serogrupy na podstawie struktury ich antygenu O – wysoce zmiennego polisacharydu na zewnętrznej błonie ich ściany komórkowej, który pozwala bakteriom unikać ochronnego działania adaptacyjnego układu odpornościowego. Naukowcy stawiają hipotezę, że antygeny O stanowią jedną z głównych przeszkód w rozpoznaniu faga. Dlatego im wyższa specyficzność faga wobec bakteryjnych antygenów O, tym skuteczniejsza jest jego terapia fagowa. Naukowcy wykorzystali kolekcję ECOR – zestaw naturalnych izolatów E. coli o różnych typach antygenów O – do przetestowania wpływu fagów Sxt1, T3 i T7 na różne szczepy. Fag Sxt1 był w stanie zainfekować 15 z 72 szczepów w kolekcji (20%), w tym wszystkie bakterie wrażliwe na T7 i/lub T3 oraz dodatkowe 7 szczepów opornych na te fagi. Następnie naukowcy przeanalizowali wpływ faga na szczep ECOR50, który jest wrażliwy na Sxt1, ale nie na T7 ani T3, i odkryli, że tylko Sxt1 był w stanie związać się z jego powierzchnią. Sugeruje to, że skuteczność Sxt1 wynika z jego zdolności do rozpoznawania receptorów bakteryjnych, a nie ze zwiększonej odporności na mechanizmy obronne komórki.
Naukowcy doszli do wniosku, że fag Sxt1, ze względu na swój rozszerzony zasięg występowania, jest skuteczniejszy niż jego krewni, fagi T3 i T7. Ma on duży potencjał terapeutyczny w leczeniu zakażeń E. coli, a ocena jego swoistości może stać się standardową procedurą charakteryzowania bakteriofagów.
W ostatnich latach, w walce z opornością na antybiotyki, trwają intensywne poszukiwania nowych bakteriofagów i opracowywanie metod ich modyfikacji. W 2021 roku amerykańscy naukowcy zintegrowali system CRISPR/Cas9 z bakteriofagiem, umożliwiając mu namierzanie określonego szczepu E. coli. W tym samym roku portugalscy naukowcy stworzyli syntetyczne bakteriofagi, które atakują Pseudomonas aeruginosa.