Amerykańscy chemicy stworzyli modele i ekspresję białek wiążących się z kompleksami porfirynowo-żelazowymi w bakteriach, a następnie wykorzystali je do katalitycznego cyklopropanowania wiązań podwójnych i wprowadzania związków diazowych do wiązań krzem-węgiel. W obu przypadkach, jak piszą chemicy w czasopiśmie „Science”, katalizatory modelowane de novo okazały się skuteczne bez użycia ewolucji kierowanej.
Reakcje cyklopropanacji i insercji do wiązań pierwiastek-wodór można przeprowadzić za pomocą katalizatorów metaloorganicznych. Zazwyczaj w takim katalizatorze metal jest związany z chiralnym ligandem, co zapewnia powstanie produktu reakcji w postaci pojedynczego izomeru optycznego. W takim podejściu wybór odpowiedniego ligandu może być trudny, a chemicy muszą testować wiele różnych katalizatorów w poszukiwaniu najskuteczniejszego.
Innym podejściem do katalizy asymetrycznej jest sprzężenie katalizatorów metaloorganicznych z białkami. W tym przypadku, zamiast chiralnych ligandów, za selektywność odpowiada struktura białka. Jeśli struktura miejsca aktywnego zostanie dobrana prawidłowo, reszty aminokwasowe będą „kierować” reakcją w miejscu aktywnym, tak aby powstał tylko jeden izomer optyczny produktu. Jeśli jednak taki katalizator białkowy nie działa prawidłowo, jego strukturę można zoptymalizować za pomocą ukierunkowanej ewolucji. Omówiliśmy, jak to działa w artykule „Zabawa w Boga”.
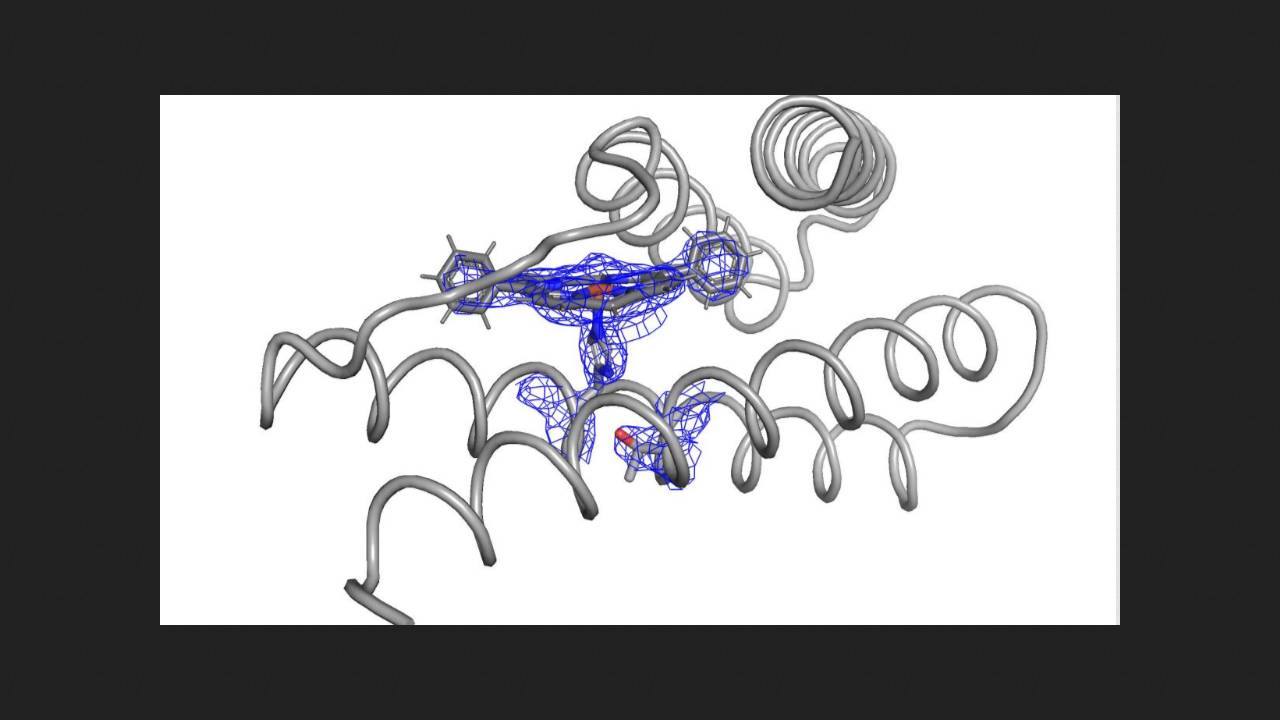
To właśnie to drugie podejście zostało niedawno zastosowane przez chemików pod kierownictwem Williama F. DeGrado z Uniwersytetu Kalifornijskiego w San Francisco. Ich pomysł polegał na modelowaniu katalizatorów białkowych dla reakcji cyklopropanacji alkenów i ich wbudowywania w wiązania krzem-wodór.
Najpierw chemicy wzięli znane białko o aktywności katalitycznej z kofaktorem difenyloporfiryną (tworzącym kompleks z jonem żelaza) i przetestowali je w reakcji cyklopropanowania styrenu diazooctanem etylu. Produkt uzyskano z wydajnością i nadmiarem enancjomerycznym wynoszącym 40%. Naukowcy postawili hipotezę, że struktura białka uniemożliwiała odczynnikom łatwą penetrację centrum aktywnego, co prowadziło do niskiej wydajności.
Aby modelować bardziej efektywne katalizatory oparte na tym białku, naukowcy wykorzystali kilka algorytmów uczenia maszynowego, w tym jeden opracowany wcześniej w ich laboratorium. W rezultacie uzyskali zbiór kilkudziesięciu struktur białkowych, z których wybrali dziesięć o najlepiej zdefiniowanych strukturach miejsc aktywnych i poddali je ekspresji w bakteriach. Większość uzyskanych białek okazała się skutecznymi katalizatorami cyklopropanacji styrenu. Najlepszym osiągniętym rezultatem była ilościowa wydajność produktu i nadmiar enancjomeryczny na poziomie około 99%.
Następnie chemicy zastosowali tę samą metodę do modelowania struktur białkowych, aby katalizować insercję związków diazowych do wiązań krzemowo-wodorowych. Tym razem jednak zamiast kofaktora difenyloporfiryny wybrali protoporfirynę IX, która działa jako prekursor hemu w komórkach. Chemicy ponownie dokonali ekspresji wybranych białek w bakteriach, co okazało się skutecznymi katalizatorami. Co więcej, naukowcy byli w stanie dodatkowo zwiększyć enancjoselektywność insercji poprzez ukierunkowaną ewolucję powstałych katalizatorów w żywych komórkach. Było to możliwe dzięki temu, że naukowcy wybrali jako kofaktor metabolit komórkowy protoporfirynę IX.
W ten sposób chemikom udało się uzyskać zestaw katalizatorów białkowych dla dwóch reakcji. Jednak pomimo wysokiej wydajności otrzymanych katalizatorów, drobne zmiany w strukturze materiałów wyjściowych często skutkowały spadkiem wydajności i enancjoselektywności.
Wcześniej informowaliśmy o tym, jak chemicy wykorzystali lipazę drożdżową w połączeniu z katalizatorem rutenowym do enancjoselektywnej syntezy makrocykli.